Normalise之后的文件用mricron查看一片白板
- Read more about Normalise之后的文件用mricron查看一片白板
- 2 comments
- Log in or register to post comments
老师们好,我做的是恒河猴的数据分析。我只有功能像的nifti数据,没有结构像的数据,可以做分析吗?
另外我Normalise之后的文件都是这样子的了(如图),这是什么原因呢。
还有根据我的情况,是否能够按照课程所说的那样处理数据呢。
老师们好,我做的是恒河猴的数据分析。我只有功能像的nifti数据,没有结构像的数据,可以做分析吗?
另外我Normalise之后的文件都是这样子的了(如图),这是什么原因呢。
还有根据我的情况,是否能够按照课程所说的那样处理数据呢。
严老师:
错误信息如图所示:
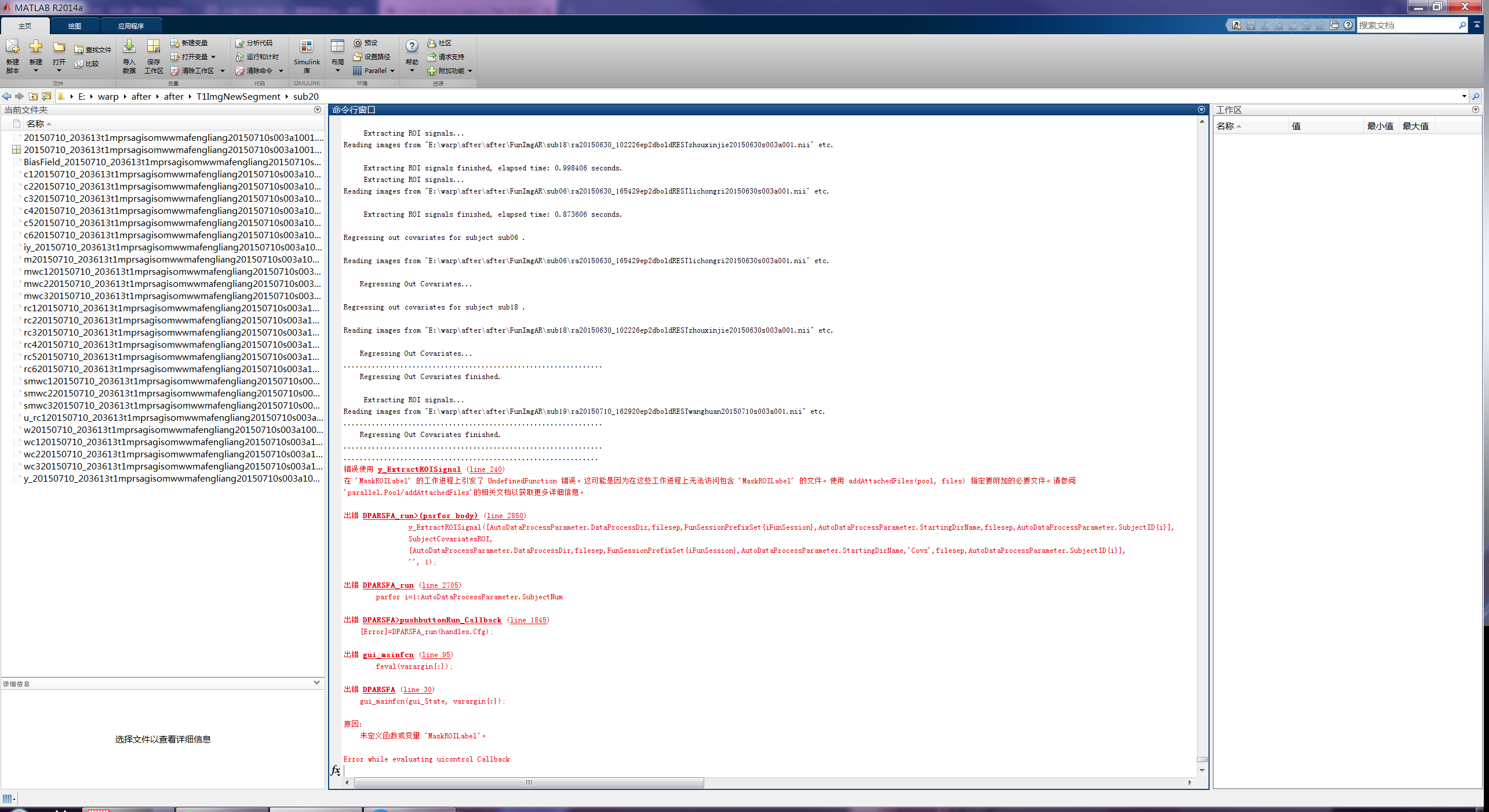
看起来是在去除协变量的时候报的错。是 y_ExtractROISignal 有bug吗?
预处理参数是默认的,没有改过
Hello, Dear experts:
I am new to fMRI analysis. I want to know, when doing the normalised procressing, i.e., warped the resting nii map into MNI space, which way the DPABI used, nonlinear warping or linear warping?
Thank you very much!
尊敬的各位老师:
您好!我想要对一个.nii文件读取它的图像矩阵,发现同一个nii文件通过y_ReadAll()与nifti工具包中的load_nii()读取出来的图像矩阵不同,有点疑惑,不知道是什么原因造成的,希望老师能给点建议。万分期待能够得到老师的回复!
尊敬的各位老师:
您好!我想要对一个.nii文件读取它的图像矩阵,发现同一个nii文件通过y_ReadAll()与nifti工具包中的load_nii()读取出来的图像矩阵不同,有点疑惑,不知道是什么原因造成的,希望老师能给点建议。万分期待能够得到老师的回复!
Dear teachers,
I performed mixed effect analysis in DPABI for my data (higher subgroup vs. lower subgroup )*two conditions (A pic vs. B pic). And I found that there is error report when I apply GRF correction for M_Interaction_F.nii created in the analysis as shown in the picture below. How to deal with it? Thank you!
Dear experts,
Thank you for creating this wonderful software. I have been finding it to be very useful!
I have a study with uneven group sizes (40 patients, 70 controls), which has caused a reviewer some concerns. My understanding is that other software packages (SPM, FSL) allow for the assumption of unequal variances to be specififed as a way to deal with this. Is this also true for DPABI, and if so which methods of correction would it apply to (GRF, permutation etc.)?
Thank you in advance,
Luke
尊敬的严老师:
我在用rest做完“two sample t-test”以后,将T图显示在了rest--slice viewer里并做了多重比较矫正,但是经过了多重比较矫正以后发现我的T图消失了,我不知道什么原因导致的,请您给予解答。
实验结果我在附件中进行了展示,请您查阅。
非常感谢。