无法使用邮箱找回密码
- Read more about 无法使用邮箱找回密码
- Log in or register to post comments
Is there any way I can make the Y-Axis list of regions completely visible? The identification of the areas is always cut and makes it difficult to identify each region without using the mouse.
This is what I'm refering to:
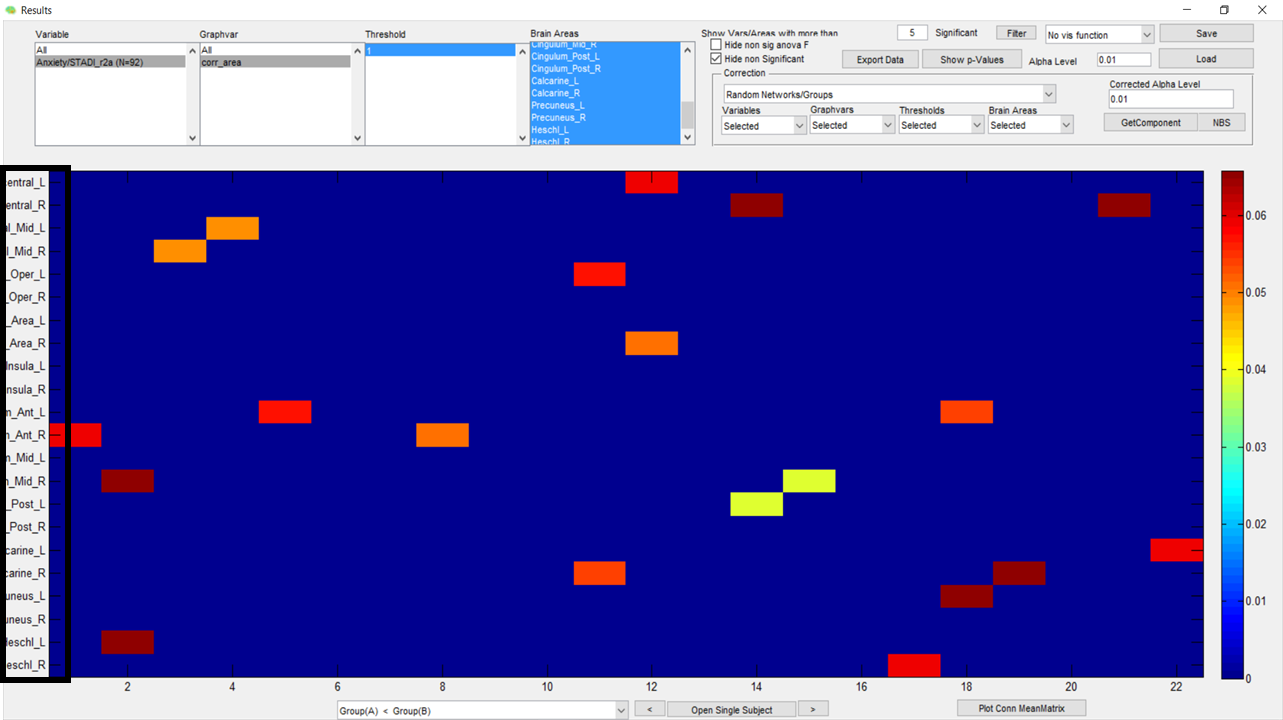
Secondly, can I alter the information on the X-Axis so that, instead of listing the areas as numericals entities only (from 1 to 22 in this case), I can identify them with their specific number on the atlas I'm using? (In this case, the AAL116)
您好!
我目前正打算使用dparsf for monkey data来预处理我的猴子大脑静息态数据。请问dparsf for monkey 与 dparsf for human有什么区别呢,dparsf for monkey data在做normalize的时候是怎么进行的呢?是默认地使用dpabi中自带的猴子模板么?另外,我在初步使用该软件的时候报了错。我的数据有6个被试,每个被试各一个session,每个session336个时间点。数据整理,我是按照 工作目录/Sub_001/FunImg/XXX.nii 和 工作目录/Sub_001/TiImg/XXX.nii 来整理的。
Matlab 2011, Win2008 , SPM8, REST Version: 1.8, Release: 20121225,
DPARSF 4.1 advanced edition
Error massage is listed below. Thank you!
Yu Bing
****************************************************************************
I'm quite new to DPARSF so I try to explain what I did so far as detailed as possible:
Hello,
I hope you can help me. Right after the step of creating the AutoMasks I get the error of
Hi,
目前我在分析数据的时候发现dparsfa里面的bet不能修改参数,部分大脑剥离的有点多,因此我自己剥离了一遍。麻烦问一下,我可以一开始在T1Img文件夹内直接存放已经剥好头骨的大脑吗,并在数据分析的时候不再勾选“bet”?会对最后结果产生影响吗?据我所知,好像剥掉头骨的大脑一般是和剥掉头骨的模板配准的,但是鉴于Dparsfa采用的时候SPM(分割以后再配准),所以不太清楚是否会有影响?另外提供剥离头骨的大脑是否会影响New segmentation进而影响配准效果呢?还望释疑问,非常感谢 :)
Hi Chao-Gan
When I ran analysis in subject space, there were some errors during betting.
What does that mean?
Thank you
Alex
-----------
Dear colleagues,