Hi all,
Besides the information provided in the Courses, there were still lots of questions about resting-state fMRI data processing. Here we'd like to build a FAQ section for your convenience. However, the FAQ is just started and will be developed gradually.
Best,
Chao-Gan
1. What are the toolboxes needed for DPABI/DPARSF?
A: DPABI/DPARSF works well with MATLAB and Statistics and Machine Learning Toolbox, Image Processing Toolbox and Signal Processing Toolbox. In addition, Parallel Computing Toolbox is needed if the users want to process multiple subjects in parallel computing mode.
2. How to solve the problem of “out of memory”?
A: Please refer to http://rfmri.org/OutOfMemory
3. What’s the ROI information of the templates provided in DPARSF?
A: Please refer to http://www.rfmri.org/node/1210#comment-1911
4. Why the data after nuisance regression looks so strange (distorted)?
A: Please refer to http://www.rfmri.org/node/1207#comment-1906
5. How to compare a single case with a group of controls?
A: Please refer to http://www.rfmri.org/node/1165
6. Does the part "Reorienting T1 Image Interactively for XXX" require any input from the user?
A: Please refer to http://www.rfmri.org/node/1082
7. What’s the input for smoothness estimation?
A: Please refer to http://www.rfmri.org/node/1118
8. What’s the option of “IsTwoTailed” in GRF correction?
A: Please refer to http://www.rfmri.org/node/1126
9. How to convert from voxel index to MNI coordinates and vice versa?
A: Please refer to http://www.rfmri.org/node/1300
10. Why linear trend should be removed before filtering?
A: Please refer to http://www.rfmri.org/node/1515
A: Please refer to http://www.rfmri.org/node/1515
11. Why "calling other os commands from MATLAB" choke the processing, e.g., dcm2nii?
A: Please refer to http://www.rfmri.org/comment/2979#comment-2979
A: Please refer to http://www.rfmri.org/comment/2979#comment-2979
12. How to solve: Error using gifti/save Too many input arguments?
Forums

TFCE correlation
老师您好:
我是在做TFCE矫正的过程中出现了以下的报错,
错误使用 palm_takeargs (line 1133)
Unknown option: "Z:\data\rest\Hippocampus_FC\dpabi_analysis\349r\TFCE\Temp\Design.csv"
出错 palm_core (line 33)
[opts,plm] = palm_takeargs(varargin{:});
出错 palm (line 81)
palm_core(varargin{:});
出错 y_GroupAnalysis_PermutationTest_Image (line 102)
palm([TempDir,filesep,'PALMConfig.txt']);
出错 y_Correlation_Image (line 74)
y_GroupAnalysis_PermutationTest_Image(DependentVolume,Regressors,OutputName,MaskFile,CovariateVolume,Contrast,'T',0,Header,PALMSettings);
出错 DPABI_STAT_TOOL>ComputeButton_Callback (line 536)
y_Correlation_Image(S, SeedSeries, OutputName, MaskFile, ImageCell, TextCell, PALMSettings);
出错 gui_mainfcn (line 95)
feval(varargin{:});
出错 DPABI_STAT_TOOL (line 42)
gui_mainfcn(gui_State, varargin{:});
出错 @(hObject,eventdata)DPABI_STAT_TOOL('ComputeButton_Callback',hObject,eventdata,guidata(hObject))
Error while evaluating uicontrol Callback
我的DPABI版本是3.1,matlab2014a,想请教一下这种报错一般是什么原因,需要怎么处理?谢谢您的回复。
看不出来是什么原因。可以试试别的电脑,或者别的数据。
看不出来是什么原因。可以试试别的电脑,或者别的数据。
谢谢严老师的指导!
谢谢严老师的指导!
你好!请问你的问题解决了吗
你好!请问你的问题解决了吗?我在校正过程中也出现了类似的问题,希望能够得到你的回复!感谢!
您好
你找到方法了吗?我也遇到了类似问题,恳请您的答复
遇到了同样的问题,同求解决方法,谢谢了
遇到了同样的问题,同求解决方法,谢谢了
<div>Chris Rorden's dcm2niiX
<div>Chris Rorden's dcm2niiX version v1.0.20200331 (JP2:OpenJPEG) (JP-LS:CharLS) MSC1900 (64-bit Windows) </div><div>Warning: only processing last of 3 input files (recompile with 'myEnableMultipleInputs' to recursively process multiple files) </div><div>Error: Input folder invalid: stimulation\FunRaw\Sub_045\GUO_QINGQING.MR.SUS_HEAD.0011.0001.2020.09.14.15.09.58.997810.96394369.IMA </div><div>Converting Functional Images:Sub_045 OK</div><div> </div><div>严老师您好!我想请教下您,我为什么用dipabi 在第一步就报错 ,dicom 文件没法转成nifti 文件 FunImg文件夹为空,请指点!</div>
Please see here to check the
Please see here to check the errors: http://rfmri.org/DPABIErrorHandling
做完AVCOVA之后在VIEWER加载结果出错
严老师您好,我做了三组数据的ANCOVA,然后用hsd做多重校正,得到的三组之间的对比,其中G1VSG2 G1VSG3,两个结果不能在VIEWER中显示,但是G2VSG3却可以正常显示,报错信息如下:
结果值里面有NaN?还是全0?
结果值里面有NaN?还是全0?
Nmin还有Pmin是0,Nmax和Pmax都有数值
Nmin还有Pmin是0,Nmax有数值,colorbar值是12.
是看这个么?
两组不能加载的Pmax是inf,能加载的那组是数值,是因为这个缘故么?
那是因为有Inf值的出现
那是因为有Inf值的出现。仔细思考一下你的图像为什么会出现Inf值。
数据整理、预处理都是按照严老师的视频来操作的
数据整理、预处理都是按照严老师的视频来操作的,还请严老师指教。谢谢!
你好,我也遇到了这个问题,请问你找到原因并解决了吗
你好,我也遇到了这个问题,请问你找到原因并解决了吗
通常可能是你的数据有些问题,想办法查找一下。
通常可能是你的数据有些问题,想办法查找一下。
可能是那两组设置有问题,结果文件为空。
可能是那两组设置有问题,结果文件为空。
repeated measures ANCOVA
严老师您好:
关于repeated measures ANCOVA在dpabi软件统计模块的使用中和ANCOVA有什么区别吗?例如只有一组病人治疗前后两个时间点的数据可以用repeated measures ANCOVA吗?我在使用这个模块时将两个时间点影像数据放进去可以出结果,但它这个和配对t检验有什么区别吗?
感谢老师
只有两个时间点用配对t检验。如果有3个时间点
只有两个时间点用配对t检验。如果有3个时间点,用repeated measures ANCOVA。
linux下dparsf运行出错
这个问题仅仅会影响pictures4checknormal
这个问题仅仅会影响pictures4checknormalization文件夹里面的图片,我的建议是无视这个问题,从FunImgARCW开始计算接下来的指标。
FieldMap Correction之后配准效果很差
严老师您好,我在对图像进行FirldMap Correction之后,图像被拉长,形状变得很奇怪,导致后面的配准效果很差,不知道这是什么原因导致的呢?以下是校正前后的影像,以及vdm5_scfpm_sc*.nii的图像:
感觉是fieldmap有问题?
感觉是fieldmap有问题?
Jial
Dear Dr Yan:
Hello!
I wanna search for help about how to adjust the order of preprocessing when preprocessing in Dpabi, for example, some projects may do Slice Timing first and then Realign, and some projects may do Realign first and then Slice Timing, and I don't see how to adjust the order of preprocessing in Dpabi, so consult you!
I wish you success in your work, happiness in your family and all the best! God bless you!
Sincerely,
Jial
Hi Jial, A simple way is to…
Hi Jial,
A simple way is to run single step for realign, starting directory name with FunImg. And then run a single step of slice timing, starting directory name with FunImgR. Then you will have FunImgRA.
您好,现在使用DPABI的时候…
您好,现在使用DPABI的时候,在matlab中输入dpabi不会出现之前的ui界面,会一直卡住在如下界面,之前使用都没问题,是最近才出现的。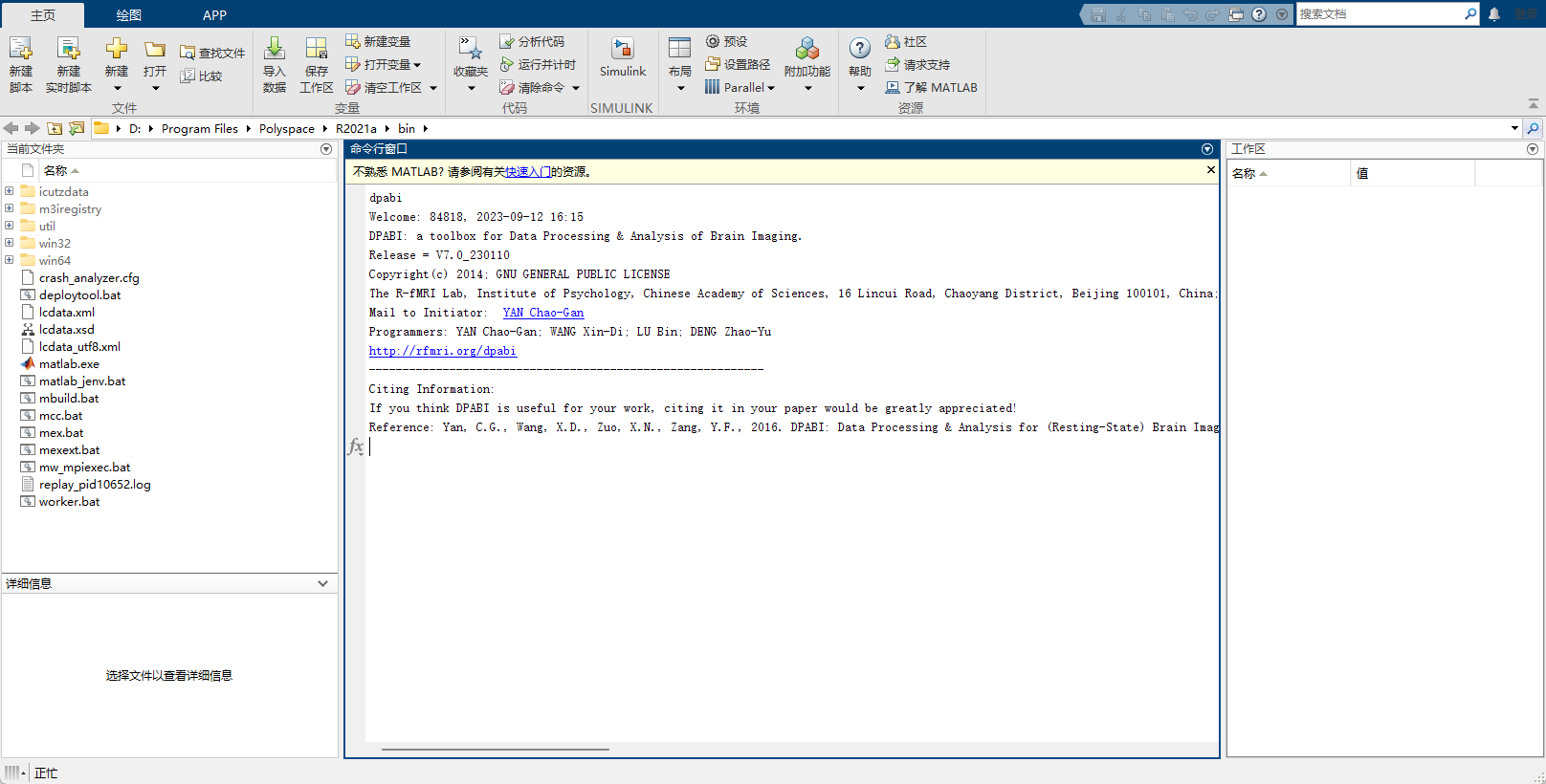 但是输入DPARSF还是可以出现ui界面的不知道是为什么,希望可以得到解答。
但是输入DPARSF还是可以出现ui界面的不知道是为什么,希望可以得到解答。
If you network is not good,…
If you network is not good, then turn off network (e.g., WIFI).
谢谢老师,解决了
谢谢老师,解决了
严老师您好! 在使用DPARSF5…
严老师您好!
在使用DPARSF5.4进行rest-fMRI预处理时normalize voxel size设置为[2 2 2](原始数据采集voxelsize=3.75,但由于fmriprep-voxelsize默认为2且很多template-voxelsize为2,所以还是设置为2进行预处理),其他参数未变,结果PicturesForChkNormalization文件夹里面所有被试的图都如下所示,但最后生成的预处理完后的.nii文件没有异常,且整个预处理过程没有报错,请问这一步标化是否存在问题?最后生成的.nii文件是否可以用于后续分析呢?
用DARTEL的话,你的数据3.75采的就不要重采样成2…
用DARTEL的话,你的数据3.75采的就不要重采样成2*2*2的了,最大3*3*3。你后面可以去reslice mask.
好的,谢谢老师!
好的,谢谢老师!
DPABIsurf报错
老师好,我用DPABIsurf处理7 T的数据,用的默认模板,没有更改参数,但是处理中报错了,希望老师能帮忙看看怎么回事?
Node: fmriprep_23_2_wf.fsdir_run_20250224_124735_393236e0_a356_42b4_ad0f_b4f9e32bc56c
Working directory: /data/fmriprepwork/sub-047TONGGUILAN/fmriprep_23_2_wf/fsdir_run_20250224_124735_393236e0_a356_42b4_ad0f_b4f9e32bc56c
Node inputs:
derivatives = /data/fmriprep
freesurfer_home = /opt/freesurfer
minimum_fs_version = 7.0.0
overwrite_fsaverage = False
spaces = ['fsaverage5', 'fsnative']
subjects_dir = /data/freesurfer
Traceback (most recent call last):
File "/opt/conda/envs/fmriprep/lib/python3.10/site-packages/nipype/pipeline/plugins/multiproc.py", line 344, in _send_procs_to_workers
self.procs[jobid].run(updatehash=updatehash)
File "/opt/conda/envs/fmriprep/lib/python3.10/site-packages/nipype/pipeline/engine/nodes.py", line 527, in run
result = self._run_interface(execute=True)
File "/opt/conda/envs/fmriprep/lib/python3.10/site-packages/nipype/pipeline/engine/nodes.py", line 645, in _run_interface
return self._run_command(execute)
File "/opt/conda/envs/fmriprep/lib/python3.10/site-packages/nipype/pipeline/engine/nodes.py", line 771, in _run_command
raise NodeExecutionError(msg)
nipype.pipeline.engine.nodes.NodeExecutionError: Exception raised while executing Node fsdir_run_20250224_124735_393236e0_a356_42b4_ad0f_b4f9e32bc56c.
Traceback:
Traceback (most recent call last):
File "/opt/conda/envs/fmriprep/lib/python3.10/site-packages/nipype/interfaces/base/core.py", line 397, in run
runtime = self._run_interface(runtime)
File "/opt/conda/envs/fmriprep/lib/python3.10/site-packages/niworkflows/interfaces/bids.py", line 960, in _run_interface
shutil.rmtree(dest)
File "/opt/conda/envs/fmriprep/lib/python3.10/shutil.py", line 725, in rmtree
_rmtree_safe_fd(fd, path, onerror)
File "/opt/conda/envs/fmriprep/lib/python3.10/shutil.py", line 664, in _rmtree_safe_fd
onerror(os.rmdir, fullname, sys.exc_info())
File "/opt/conda/envs/fmriprep/lib/python3.10/shutil.py", line 662, in _rmtree_safe_fd
os.rmdir(entry.name, dir_fd=topfd)
OSError: [Errno 39] Directory not empty: 'label'