使用最新版本DPABI/DPARSF做预处理时遇到如下报错信息
- Read more about 使用最新版本DPABI/DPARSF做预处理时遇到如下报错信息
- 4 comments
- Log in or register to post comments
严老师:
错误信息如图所示:
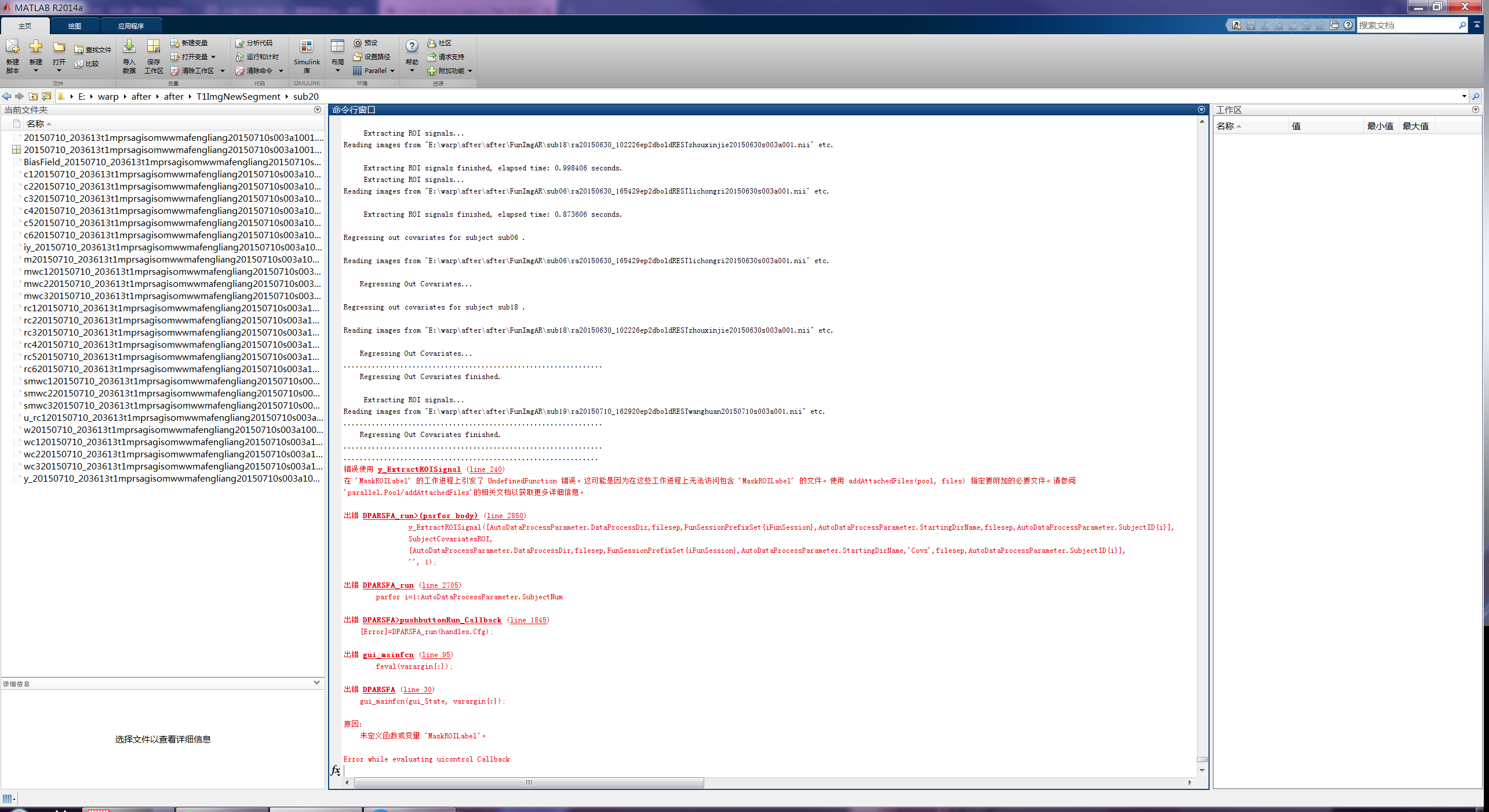
看起来是在去除协变量的时候报的错。是 y_ExtractROISignal 有bug吗?
预处理参数是默认的,没有改过
采用我们的母语,能够进行迅捷有效的沟通。但是鉴于英文是科技通用语言,建议所有研究者采用英文发贴,做为训练自己英语的良机。如果你实在觉得英文无法表达你的思想,请在该论坛中采用美丽的中文提问。
严老师:
错误信息如图所示:

看起来是在去除协变量的时候报的错。是 y_ExtractROISignal 有bug吗?
预处理参数是默认的,没有改过
尊敬的各位老师:
您好!我想要对一个.nii文件读取它的图像矩阵,发现同一个nii文件通过y_ReadAll()与nifti工具包中的load_nii()读取出来的图像矩阵不同,有点疑惑,不知道是什么原因造成的,希望老师能给点建议。万分期待能够得到老师的回复!
尊敬的各位老师:
您好!我想要对一个.nii文件读取它的图像矩阵,发现同一个nii文件通过y_ReadAll()与nifti工具包中的load_nii()读取出来的图像矩阵不同,有点疑惑,不知道是什么原因造成的,希望老师能给点建议。万分期待能够得到老师的回复!
尊敬的严老师:
我在用rest做完“two sample t-test”以后,将T图显示在了rest--slice viewer里并做了多重比较矫正,但是经过了多重比较矫正以后发现我的T图消失了,我不知道什么原因导致的,请您给予解答。
实验结果我在附件中进行了展示,请您查阅。
非常感谢。
尊敬的老师:
我在用rest的slice viewer去查看two sample t-test的结果时,我加入的T图(overlay)和我的底图(underlay)不能对齐。我不太清楚是什么原因导致的,希望您能给予解答,不甚感激!
我附上了我的结果,麻烦查阅一下。
大家好,最近准备换电脑,想请教一下有人的电脑是Ryzen 4000系列的处理器的吗?目前和matlab以及DEPABI的兼容性怎么样?十分感谢!
尊敬的严教授,您好,我想请教一下关于求灰质体积的问题:
我们在求海马的体积的时候,用您的软件:DPABI_ROISignalExtracter.m这个工具的时候,实际上是求的这个mask内所有体素的mean值,如果改成求mask内的所有体素的和(sum)可以吗?
这两个有区别吗,谢谢老师!
老师,我想请问一下,怎么把AAL3模板加载到dpabi里,点击cluster report就可以根据AAL3模板来报脑区即坐标?谢谢
严老师,
您好!我做了一组人群基于种子点的FC的单样本T,想用单样本T的结果做一个mask,限制后面的比较次数,想请问您怎样用单样本T的结果做mask呢?是应该做一个0,1的二值mask吗?希望您有空回复一下,谢谢老师呀!
严老师,
您好!我做了一组人群基于种子点的FC的单样本T,想用单样本T的结果做一个mask,限制后面的比较次数,想请问您怎样用单样本T的结果做mask呢?是应该做一个0,1的二值mask吗?希望您有空回复一下,谢谢老师呀!