静息态Normalize报错!
- Read more about 静息态Normalize报错!
- 1 comment
- Log in or register to post comments
严老师:
您好,我这两天在做静息态预处理时,Normalize无论选择by using EPI templates 还是by DARTEL时,都会报错,不知道是什么原因,希望老师能够帮我找找原因。设置参数和错误代码如图所示,使用的是DPABI_5.1 和SPM12b。
采用我们的母语,能够进行迅捷有效的沟通。但是鉴于英文是科技通用语言,建议所有研究者采用英文发贴,做为训练自己英语的良机。如果你实在觉得英文无法表达你的思想,请在该论坛中采用美丽的中文提问。
严老师:
您好,我这两天在做静息态预处理时,Normalize无论选择by using EPI templates 还是by DARTEL时,都会报错,不知道是什么原因,希望老师能够帮我找找原因。设置参数和错误代码如图所示,使用的是DPABI_5.1 和SPM12b。
严老师:
您好,我这两天在做静息态预处理时,Normalize无论选择by using EPI templates 还是by DARTEL时,都会报错,不知道是什么原因,希望老师能够帮我找找原因。设置参数和错误代码如图所示,使用的是DPABI_5.1 和SPM12b。
严老师:
您好!
我用DPABI统计工具箱中的ANCOVA做三组间的方差分析,并选用tukey-Kramer进行post-hoc分析。之后再对两组间的统计结果(比如F_PariwiseDiff_Z_G1vsG2.nii)进行多重比较校正以观察两组间的统计显著区域,在多重比较校正阶段,我对两组间的Z图选用GRF(位于DPABI_VIEW工具中),但发现无法正确估计FWHMx/y/z以及dLh,这些指标估计出来都是Nan,前面ANCOVA的结果也是利用DPABI计算的。请问老师这样操作是否有问题以及有何建议?
谢谢老师!
老师您好,我已经run好几个小时了,没有报错,但是并没有新的文件生成,每完成一个预处理过程都要生成新的文件吗??MATLAB也没有新信息出现,请问是什么原因呢??
严老师:
您好!
我根据DPABI教程计算多组被试之间的影像差异后,想进一步分析多组被试间的临床信息是否具有差异,比如某一症状,但在这个过程中也需要回归站点、性别、年龄等因素对这个症状的影响,请问老师DPABI可以处理这种临床信息的anova分析吗,或者老师有没有可用的工具推荐?
此外,这对这个问题,我查阅了一些资料,发现协变量的顺序可能也会影响最终的结果,对于这一问题老师有什么建议吗?
期待老师的回复!
谢谢老师!
各位老师好,我在做恒河猴数据分析的时候,做出来的groupmask不太匹配得上我的脑部,所以计算指标的时候就会出现图中显示的那种情况,有一些脑范围之外的数值。我可以每个猴脑独立抠一个自己的mask,独立计算它的那些指标,最后再一起分析吗。是哪一步出了问题吗,这种情况我应该怎么解决呢。感谢!
尊敬的各位老师:
您好!关于皮层预处理时我有些疑问,我想问下dpabisurfer在进行预处理的时候有没有进行realign,realign这一步骤是不是相当于fmriprep的功能预处理那里的generate reference&brain mask 那一步?万分期待老师您的回复!
严老师:
我从FunImgARCWF目录开始,计算VMHC,错误信息和DPARSF设置如下:
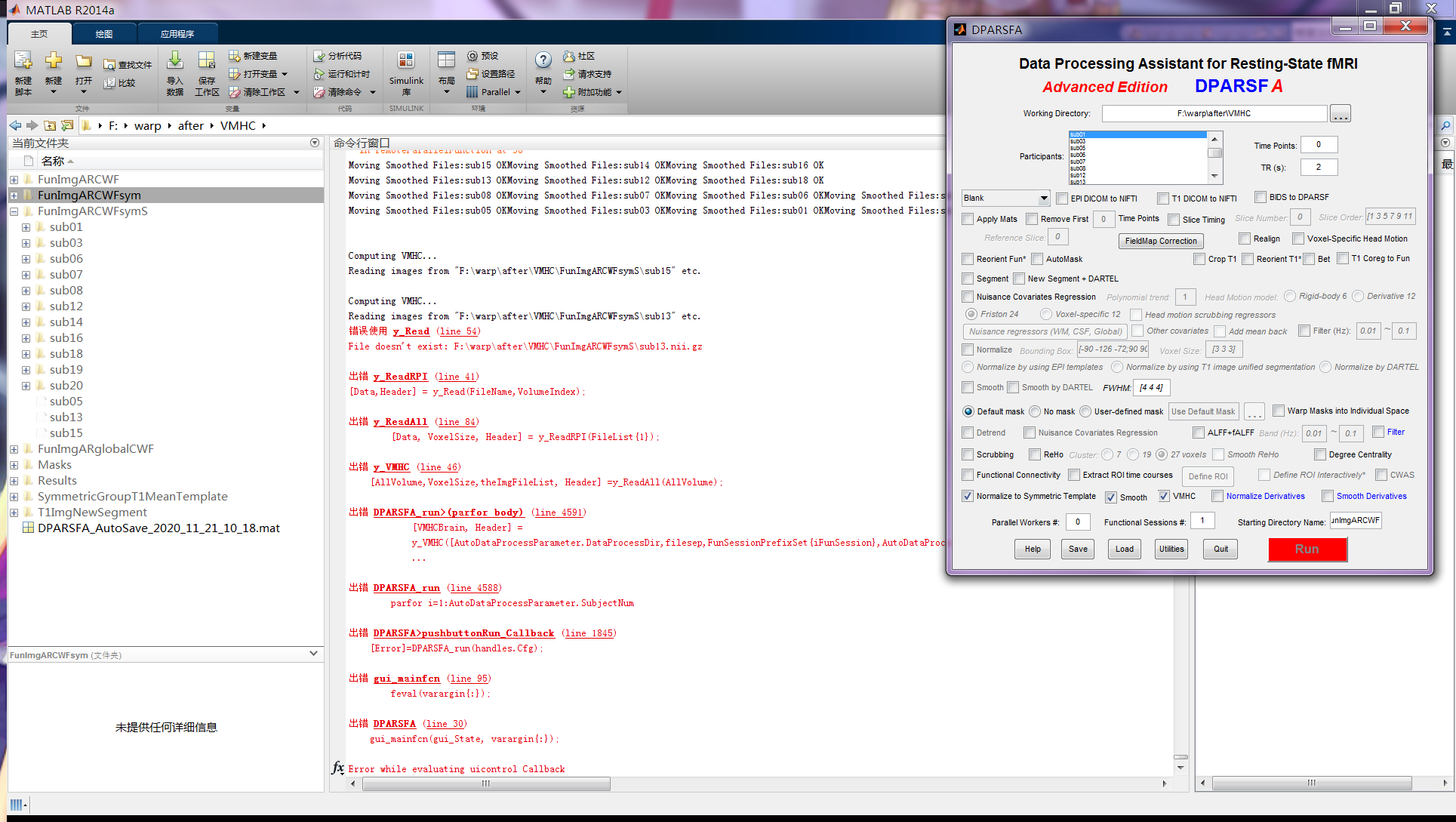
这是数据质量的问题吗?
老师们好,我做的是恒河猴的数据分析。我只有功能像的nifti数据,没有结构像的数据,可以做分析吗?
另外我Normalise之后的文件都是这样子的了(如图),这是什么原因呢。
还有根据我的情况,是否能够按照课程所说的那样处理数据呢。