DPABISurf 处理过程中 报错求助 [GIFTI]
- Read more about DPABISurf 处理过程中 报错求助 [GIFTI]
- 1 comment
- Log in or register to post comments
采用我们的母语,能够进行迅捷有效的沟通。但是鉴于英文是科技通用语言,建议所有研究者采用英文发贴,做为训练自己英语的良机。如果你实在觉得英文无法表达你的思想,请在该论坛中采用美丽的中文提问。
严老师您好!
我使用自己的数据跑DPABISurf Pipeline提取了时间序列,然后导入提取的时间序列到DPABINet做网络构建时出现如下报错,Network Construction设置也在图中,
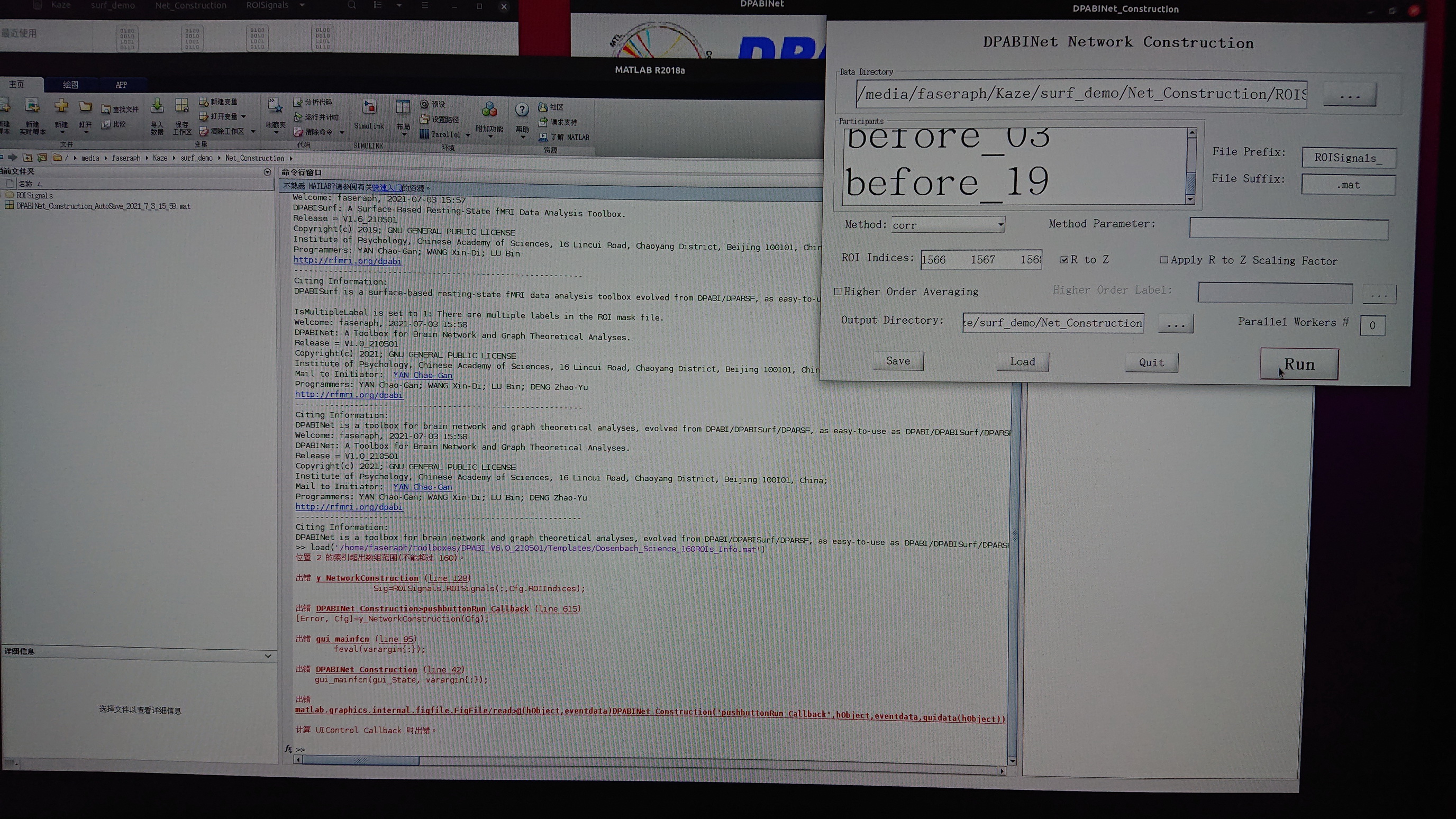
我又尝试了用DPARSF预处理时提取的时间序列做网络构建,还是一样的报错信息,预处理Pipeline和ROI List如下图所示,

请问是网络构建设置有误,还是提取时间序列的设置有误?
尊敬的严老师好:
我在使用restmetaMDD数据集,想用dpaBI做功能连接、rest做效应连接。查阅论坛,以及详细数据结构、批处理管道描述,发现文件结构ROISingals_FunImgARCWF,应该未经过平滑。
各位专家,想请教一下,最近在做不同亚型精神障碍皮层特征分析,未经过TFCE检验的只有很少结果能够通过FDR或FWE校正,但是经过TFCE检验后有更多结果能够通过FDR或FWE校正,但结果与未经过TFCE检验的结果不一致,这种情况有可能发生吗?为什么会出现这种情况?感觉过程中步骤没错,但第一次用TFCE检验,只理解这是一种非参数检验,也没查到更深入的资料,担心这种情况的发生会不会结果不可靠,还望高人指点!
尊敬的严教授,我在用您的软件出来您网站上的DEMO数据的时候报错,见文件
请问有啥解决方案吗,谢谢
严老师好:
我对ADNI上下载的原始dicom文件进行预处理,将其转为nii格式后出现两种状态,如下图所示,1:正常的为上方所示,2:异常的如下方所示,各voxel的值过大,造成后续预处理有问题,请问原因是什么?该如何解决?
谢谢老师
严老师您好:
我使用ADNI公开数据下载后原始dcm图像后进行ARWS的预处理,在检查处理后的图像数据时发现,个别被试出现如图片所示情况,请问这是由什么原因导致?
谢谢老师!
老师,您好,我想问一下我在使用DPARSF进行预处理及ALFF,ReHo以及FC等特征提取时是选择在所有特征计算完后进行smooth derivatives,这时候ROI-wise的FC的文件夹是ROIsignals_FunImgARCWF是基于没有进行平滑处理的数据计算的,请问这时候得到的ROI-wise的FC可以直接使用进行下一步分析么?还是说需要在预处理后的数据基础上再进行smooth后再计算ROI-wise的FC呢?
严老师你好,我在跑完BET之后,做T1的Segment,出现了如下报错,请问如何解决?